ここでは NCBI Home Page (http://www.ncbi.nlm.nih.gov/) を利用して,DNA配列データをダウンロードする.なお,以下の説明は2002年9月に作成したもので,現在ではNCBIのウェブの構成が若干異なっている.例えばデータベースに登録されているゲノム情報は日々増加していることを反映して,表示される登録数は増えている.
あらゆる生物のDNA配列が公共のデータベースに登録されている.ヒトのミトコンドリアDNAの情報を取り出す様子を以下に示す.ミトコンドリアは細胞の小器官であるが独自のDNA(mtDNA)を持ち,細胞の核DNAとは独立に増殖する.一細胞あたり数百から数千のミトコンドリアが存在する.ミトコンドリアDNAは進化系統樹推定によく用いられている.
では NCBI Home Page (http://www.ncbi.nlm.nih.gov/)をアクセスしてみよう.つぎのようなページが見えるだろう.
左のバーにある"Molecular databases"をクリックすると次のページになる.
NCBI Databases (http://www.ncbi.nlm.nih.gov/Database/index.html)
左のバーにある"Taxonomy"をクリックすると,生物の分類のページになる.そこから "Eutheria"(胎盤をもつ哺乳類)までたどると,次のページになる.
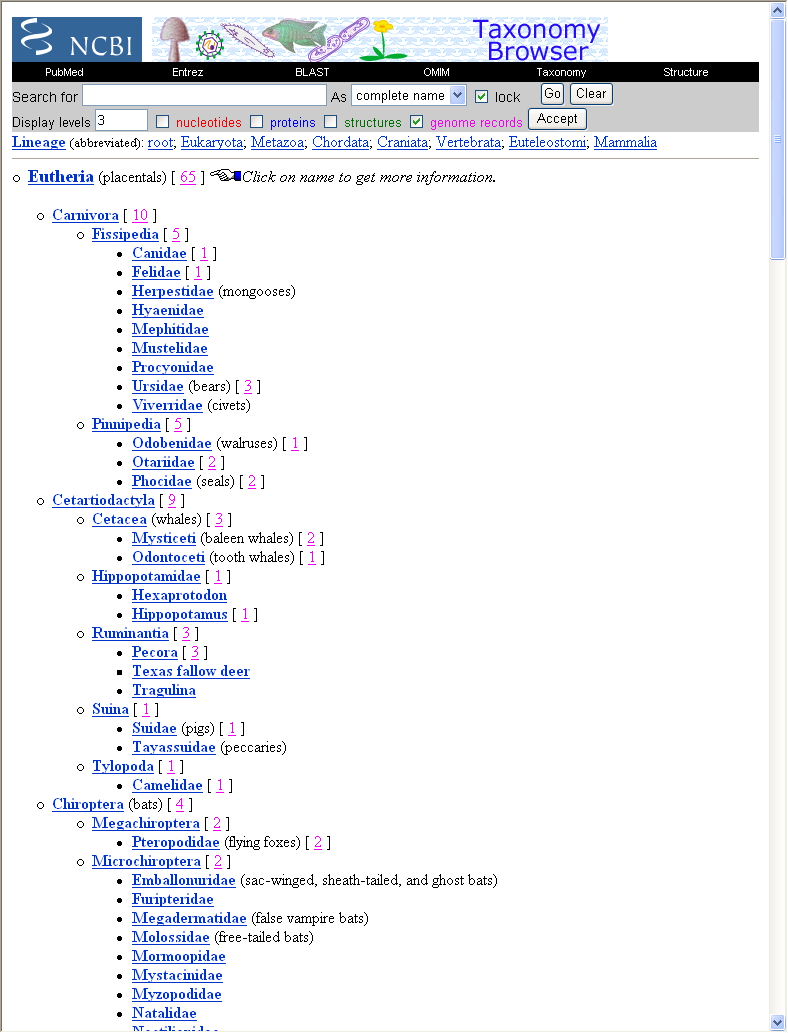
ここでは"genome records" (ゲノム レコード)にチェックマークをつけてあるので,complete genomeが登録されている数が分類群ごとに示される.つまりゲノムが完全に解読されているものだけを表示する.これは比較的少数であるが,ここではこれを用いる(くわしくは,ここを参照).もし,"nucleotides"にチェックマークを付けると部分的にしか得られていない塩基配列もすべて表示されるので,その数は莫大になる.
ヒトの核には1番から22番までの染色体が2本づつあり,さらに性別によってX染色体やY染色体がある.全ゲノムのサイズは約3G塩基対(A,T,G,Cの並びが3,000,000,000程度の長さ)である.これにくわえてミトコンドリアにもゲノムがあり,サイズは16K塩基対(16,000程度の長さ)である.われわれの分析では,ミトコンドリア(単数形itochondrion)t攀ゲノムだけを分析に用いる.

ここで見ている Eutheria に含まれる生物群のリストが一覧に(階層的に)示されている.基本的には学名で表示されているが,カッコ内に通称名 (common name) も示されているものもある.一方,共通祖先に対応する系統樹の根(root)からutheriakご?6階層的な分類群は "lineage"のところに示されている.ここで例えば "Vertebrata"をクリックするとEutheriaの上位概念である脊椎動物へジャンプする.
Eutheriaのページを少し下のほうに見て行くと次のように"Primates" (霊長類)があるので,それをクリックしよう.

Primatesもまた次のように階層的になっている.ゴリラ,ヒト,チンパンジーなどを含む生物群が"Hominidae"である.このなかの"Homo"がヒトに対応する.これをクリックしよう.

Homoの "Homo sapiens"が現代人である.とくに"Homo sapiens neanderthalensis"はネアンデルタール人である.Homo sapiensの横には,このgenome recordが1件あることがカッコ内の数値で示されている.これをクリックしよう.

ようやくデータベースのエントリーにたどり着いた.NC_001807はこのエントリーの受け入れ番号 (accessoion number)である.その下の"Homo sapiens mitochondrion"はヒトミトコンドリア,"complete genome"はその全ゲノムであることをあらわす.

このアクセッションナンバーはNCで始まるが,これはNCBIで準備している標準シーケンスであることをあらわす.(下のほうに"ref"とあるのも同じことをあらわしている.) つまり,ヒトのミトコンドリアゲノムといっても色々あって個体差があるはずだが,その代表をひとつ選んで登録しているわけである.もし,色々なヒトのミトコンドリアゲノムを入手したければここをクリックしてみよ.いろいろな地域に住むヒトのミトコンドリアゲノムから推定した系統樹は,たとえば次のようになる.
また,色々な生物のミトコンドリアゲノム一覧をまとめてみたければ,ここをクリックしてみよ.例えば哺乳類の系統樹を推定すると次のようになる.(上側は形態学から推定した系統樹,下側はDNAから推定した系統樹)
もし情報のアクセッションナンバーがはじめから分かっていれば,NCBIのホームページのSearchで直接入力すればよい.
NC_001807のエントリーをクリックするとヒトのミトコンドリアゲノムがグラフィックで表示される.
Homo sapiens mitochondrion, complete genome
http://www.ncbi.nlm.nih.gov/cgi-bin/Entrez/framik?db=genome&gi=12188

ミトコンドリアゲノムは環状で,DNAの長さは16kbp程度 (16000 bp,つまり16000個のA,T,G,Cの並びということ)と,非常にコンパクトである.ヒトの核DNAが数Gbpであることと比べると大変小さい.上のグラフィックスからも読み取れるようにミトコンドリアDNAはいくつかの要素から構成されている.ND1, ND2, COX1, COX2, ATP8, ATP6, COX3, ND3, ND4L, ND4, ND5, ND6, CYTBとい表示されているのは,13個のたんぱく質をコードしている遺伝子である.tRNAはトランスファーRNAと呼ばれるもので,DNAの並びをアミノ酸に変換するときの変換表の情報を持っている.rRNAはリボゾームRNAであり,遺伝子のDNA情報をたんぱく質に変換する.あえて分かりやすくミトコンドリアゲノムを「自己解凍ファイル」にたとえると,解凍プログラムはrRNAとtRNAから構成されていてrRNAがコード領域,tRNAがデータ領域である.そして解凍されるプログラム本体が13個の遺伝子に対応する.
13個の遺伝子の部分ではDNA配列は3個ごとに読み出されてアミノ酸に変換される.たんぱく質というのはこのアミノ酸の並びのことである.この3個の組は「コドン」と呼ばれる.DNAのアルファベットはA,T,G,Cの4個であるからその3個組は4の3乗で64通りである.一方変換されるアミノ酸は20個のアルファベットである.読み出し開始コドンやストップコドンを含めてもこの符号は冗長である.コドンからアミノ酸への変換表は脊椎動物のミトコンドリアの場合はコドン変換テーブル2(またはNCBIのテーブル一覧)である.この変換の様子をグラフィックスで見たければ,以下のページで"Display"横のタブを"Graphics"にした上で"Display"をクリックせよ.

さて,NC_001807をクリックしてデータエントリ本体を見よう.
http://www.ncbi.nlm.nih.gov/entrez/viewer.fcgi?val=NC_001807

これを保存するには,"Display"横の"Text"をクリックしてからブラウザの「名前を付けて保存」や「Save as」の機能を使ってテキスト形式でファイルを保存する.ファイル名はアクセッション番号 NC_001807.txt としておく.
同様にして自分の興味のある他の生物のファイルも作成する.とりあえず脊椎動物のミトコンドリアゲノムだけに限定しておくことを薦める.それ以外では「変換表」も異なってきて面倒である.
ダウンロードしたデータファイルはGenBank Flat File Format (http://www.ncbi.nlm.nih.gov/Sitemap/samplerecord.html)にサンプルを使って読み方が解説されている.とりあえず我々はたんぱく質をコードしている13個の遺伝子しか解析の用いないので,「CDS]と書かれたレコードだけわかればよい.上図では,CDS 3308..4264 /gene="ND1"とあるが,これはND1の遺伝子がゲノムの3308番から4264番までにあることを意味している.変換されたアミノ酸配列は/translationのところにある.ファイルの一番最後にORIGINから始まるa,t,g,cのアルファベットの並びがあるが,これがゲノムである.したがって,この並びの3308..4264を取り出してきて,コドン変換表2を適用すると先ほどの/translationの内容が得られるはずである.